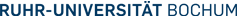PD Dr. Markus Piotrowski
Ars legendi Fakultätenpreis
 Herzlichen Glückwunsch an Markus Piotrowski von unserem Lehrstuhl zum Gewinn des »Ars legendi Fakultätenpreis« für exzellente Hochschullehre (Biologie) 2019!
Herzlichen Glückwunsch an Markus Piotrowski von unserem Lehrstuhl zum Gewinn des »Ars legendi Fakultätenpreis« für exzellente Hochschullehre (Biologie) 2019!
Zur Stifterverband-Webseite | Presseerklärung RUB
Biosynthesis of the metal chelator molecule nicotianamine
(nur englisch)
Background and preliminary work: The Krämer group has discovered that a plant membrane transport protein, Arabidopsis thaliana Zinc-Induced Facilitator 1 (ZIF1), mediates the vacuolar sequestration of nicotianamine (NA). NA is a non-proteinogenic amino acid that acts as a high-affinity metal chelator molecule in metal homeostasis of plants and filamentous fungi. Furthermore, NA has promising anti-hypertensive pharmacological activity. Roots of Arabidopsis thaliana overexpressing AtZIF1 accumulate ~30-fold enhanced NA concentrations. Nicotianamine synthases (NAS) of plants and filamentous fungi convert three S-adenosylmethionine (SAM) molecules to one NA molecule. NAS catalyses several consecutive reactions in a single active site, as concluded based on crystal structures of an NAS-like archaeal thermoNAS. Its reaction product, thermoNA, contains a glutamate instead of the terminal SAM-derived unique azetidine-2-carboxylate moiety that is characteristic of NA.
NAS proteins were first identified in barley and other plant species, and only later in filamentous fungi. More recently, NAS-like genes were found to be part of a Pseudomonas aeruginosa operon essential for survival on human airway mucus secretions, and to act in the synthesis of an NA-related compound of Staphylococcus aureus that utilizes only a single SAM molecule. Interestingly, no proteins acting in the catabolism of NA are known. A possible NA breakdown product – the toxic proline analogue and allelopathic chemical azetidine-2-carboxylate – was detected in only a few plants to date.
CN-Hydrolasen Höherer Pflanzen
Enzyme sind Biokatalysatoren, die den Ablauf einer chemischen Reaktion beschleunigen. Somit können Reaktionen, die sonst nur unter extremen Bedingungen (z.B. hohe Temperaturen oder Drücke, extreme pH-Werte) stattfinden, unter „normalen“ Bedingungen, wie sie in einer Zelle vorzufinden sind, durchgeführt werden. Die C-N-Hydrolasen, an denen wir arbeiten, sind Enzyme, die, wie der Name schon ausdrückt, C-N-Bindungen hydrolytisch spalten (EC. 3.5.x.x: Hydrolasen, die C-N-Bindungen, aber nicht Peptidbindungen spalten).
![]() Alleine in dem kleinen Genom der Ackerschmalwand Arabidopsis thaliana sind 9 Gene für verschiedene C-N-Hydrolasen vorhanden. Wir untersuchen zurzeit schwerpunktmäßig die Nitrilasen (4 Gene in A. thaliana) und die Beteiligung der C-N-Hydrolasen an der Polyaminbiosynthese.
Alleine in dem kleinen Genom der Ackerschmalwand Arabidopsis thaliana sind 9 Gene für verschiedene C-N-Hydrolasen vorhanden. Wir untersuchen zurzeit schwerpunktmäßig die Nitrilasen (4 Gene in A. thaliana) und die Beteiligung der C-N-Hydrolasen an der Polyaminbiosynthese.
1. Nitrilasen
Nitrilasen (EC 3.5.5.x) katalysieren die Umsetzung von Nitrilen (= Cyanoverbindungen: R-C≡N) zu den entsprechenden Carbonsäuren. Die vier Nitrilasen von A. thaliana (NIT1-NIT4) lassen sich sowohl aufgrund der Sequenzähnlichkeiten untereinander als auch aufgrund der Substrate, die sie umsetzen können, in zwei Gruppen aufteilen: NIT1-NIT3 (NIT1-Gruppe) und NIT4.
1.1. Evolution der Nitrilasen in glucosinolatbildenden Pflanzen
Nitrilasen vom Typ der NIT1-Gruppe sind bisher nur von wenigen Vertretern der Pflanzenfamilie der Kreuzblütler (Brassicaceen) bekannt, so z.B. in A. thaliana, Raps und Kohl. Diese Pflanzenfamilie ist gleichzeitig für das Vorhandensein von so genannten Glucosinolaten (Senfölglycosiden) bekannt. Bei Verletzung der Pflanze – z.B. durch den Biss eines pflanzenfressenden Insektes – werden Enzyme freigesetzt, die aus den Glucosinolaten Isothiocyanate, Thiocyanate und Nitrile freisetzen, die der Abwehr des Fraßfeindes dienen sollen (Abbildung 1). In A. thaliana sind über 30 verschiedene solcher Glucosinolate bekannt, und dementsprechend vielfältig ist auch das Spektrum an Nitrilen, das daraus entstehen kann. Das passt gut mit der Tatsache zusammen, dass die Nitrilasen der NIT1-Gruppe sehr unterschiedliche Nitrile umsetzen können.
![]() In verschiedenen Pflanzenarten gibt es aber auch unterschiedliche Glucosinolate. Wir untersuchen daher, ob sich die verschiedenen Nitrilasen der NIT1-Gruppe an die spezifische Glucosinolatzusammensetzung ihrer Pflanzen angepasst haben, und wie diese Anpassung auf molekularer Ebene vollzogen wurde (welche Veränderungen der Enzyme haben stattgefunden?).
In verschiedenen Pflanzenarten gibt es aber auch unterschiedliche Glucosinolate. Wir untersuchen daher, ob sich die verschiedenen Nitrilasen der NIT1-Gruppe an die spezifische Glucosinolatzusammensetzung ihrer Pflanzen angepasst haben, und wie diese Anpassung auf molekularer Ebene vollzogen wurde (welche Veränderungen der Enzyme haben stattgefunden?).
![]() Ein wichtiger Aspekt im Zusammenhang des Glucosinolat/Nitrilase-Systems ist die Tatsache, dass aus einem bestimmten Glucosinolat – dem Glucobrassicin – Indol-3-acetonitril freigesetzt werden kann. Indol-3-acetonitril kann in vitro durch die Arabidopsis-Nitrilasen NIT1, NIT2 und NIT3 zu dem pflanzlichen Wachstumshormon Indol-3-essigsäure umgesetzt werden (Abbildung 1). Ob Nitrilasen in vivo (in der Zelle) tatsächlich an der Indol-3-essigsäure-Biosynthese beteiligt sind, steht noch offen.
Ein wichtiger Aspekt im Zusammenhang des Glucosinolat/Nitrilase-Systems ist die Tatsache, dass aus einem bestimmten Glucosinolat – dem Glucobrassicin – Indol-3-acetonitril freigesetzt werden kann. Indol-3-acetonitril kann in vitro durch die Arabidopsis-Nitrilasen NIT1, NIT2 und NIT3 zu dem pflanzlichen Wachstumshormon Indol-3-essigsäure umgesetzt werden (Abbildung 1). Ob Nitrilasen in vivo (in der Zelle) tatsächlich an der Indol-3-essigsäure-Biosynthese beteiligt sind, steht noch offen.
1.2. Beteiligung von Nitrilasen an der Cyanidentgiftung
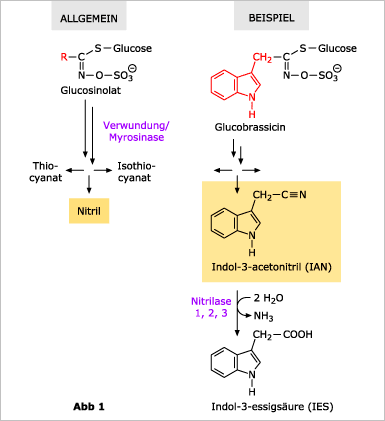
Pflanzen können Cyanid (HCN = Blausäure) entgiften, indem sie dieses an eine Aminosäure koppeln (siehe Abbildung 2). Das entstehende 3-Cyano-L-alanin (kurz: Cyanoalanin) ist für die Pflanze jedoch „wertlos“ und wird daher zu Asparagin, einer für die Pflanze „nützlichen“ Aminosäure, umgesetzt. Neben der Entgiftung wird somit auch der im Cyanid enthaltene Stickstoff zurückgewonnen. Die Arabidopsis-Nitrilase NIT4 kann Cyanoalanin sowohl zu Asparaginsäure (eine ebenso „nützliche“ Aminosäure) als auch zu Asparagin umsetzen. Das besondere an NIT4 ist, dass dieses Enzym, im Gegensatz zu den Nitrilasen NIT1-NIT3 (die auf eine Pflanzenfamilie beschränkt zu sein scheinen), in sehr vielen Pflanzen zu finden ist. Unsere aktuellen Untersuchungen zielen darauf ab zu prüfen, inwieweit NIT4 tatsächlich an der Cyanidentgiftung und am Stickstoff"recycling" in der Pflanze beteiligt ist.
2. Biosynthese des Polyamins Putrescin in Pflanzen
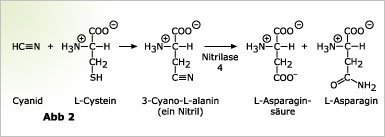
Der Begriff „Polyamin” ist etwas irreführend, tatsächlich meint man damit ganz bestimmte kleine Oligoamine, die in allen lebenden Organismen gefunden werden. Obwohl die genaue Funktion dieser Moleküle in den allermeisten Fällen nicht verstanden ist, weiß man, dass sie an einer Reihe physiologischer Funktionen beteiligt sind. In Pflanzen wird Polyaminen unter anderem eine wichtige Rolle bei der Zellteilung und bei der Antwort auf Salzstress zugesprochen. Das einfachste Polyamin, was aber auch als Vorstufe für weitere Polyamine dient, ist das Putrescin. Putrescin kann entweder aus den Aminosäuren Ornithin oder Arginin gebildet werden. Die Biosynthese des Putrescins aus Arginin findet man im Wesentlichen bei Pflanzen und bestimmten Bakterien. Den abschließenden Schritt in diesem Biosyntheseweg katalysiert die N-Carbamylputrescin-Amidohydrolase (kurz: CPA), die N-Carbamylputrescin zu Putrescin umsetzt (Abbildung 3). Diese CPA gehört zu der Familie der C-N-Hydrolasen.
![]() Ein weiteres Enzym in diesem Stoffwechselweg, die Agmatin-Iminohydrolase (kurz: AIH), katalysiert ebenfalls eine C-N-Hydrolase-Reaktion, es gehört aber nicht zur Familie der C-N-Hydrolasen, da es mit diesen nicht verwandt ist.
Ein weiteres Enzym in diesem Stoffwechselweg, die Agmatin-Iminohydrolase (kurz: AIH), katalysiert ebenfalls eine C-N-Hydrolase-Reaktion, es gehört aber nicht zur Familie der C-N-Hydrolasen, da es mit diesen nicht verwandt ist.
![]() Nachdem wir diese beiden Enzyme von A. thaliana kloniert und ihre Funktion gezeigt haben, beschäftigen wir uns aktuell mit den entsprechenden Enzymen anderer Organismen.
Nachdem wir diese beiden Enzyme von A. thaliana kloniert und ihre Funktion gezeigt haben, beschäftigen wir uns aktuell mit den entsprechenden Enzymen anderer Organismen.
Weitere Informationen
Wenn Sie an weiteren Informationen interessiert sind, besuchen Sie bitte meine "C-N-Hydrolase Group"-Homepage.
![]()