Molekulare und funktionelle Charakterisierung pflanzlicher Glutamatrezeptorhomologe
Ionotrope Glutamatrezeptoren sind in Tieren bereits seit vielen Jahrzehnten bekannt. Im Gehirn von Wirbeltieren spielen diese ligandenaktivierten Ionenkanäle eine entscheidende Rolle in der Kommunikation zwischen Nervenzellen. Die Senderzelle schüttet dabei an der Kontaktstelle mit der Empfängerzelle den Botenstoff Glutamat aus. Der aktiviert an der Oberfläche der Empfängerzelle Glutamatrezeptoren, die daraufhin ihren eingebauten Kanal öffnen und so Ionen in die Zelle strömen lassen. Das erzeugt ein elektrisches Signal, das anschließend in der Empfängerzelle weitergeleitet wird.
 Im Jahr 1998 entdeckte man auch in der Pflanze Arabidopsis thaliana mögliche Glutamatrezeptorgene. Das war ein überraschender Fund, weil Pflanzen kein Nervensystem oder gar Gehirn haben. Dennoch besitzt Arabidopsis sogar zwei Glutamatrezeptorgene mehr als Wirbeltiere, wie sich nach der Sequenzierung ihres Erbgutes im Jahr 2000 herausstellte. Die Proteine, die von diesen Genen kodiert und als GLRs (glutamate-like receptors, glutamatähnliche Rezeptoren) bezeichnet werden, ähneln den Glutamatrezeptoren der Tiere sowohl in ihrer Sequenz als auch in ihrer vorhergesagten Struktur. Das legte die Vermutung nahe, daß auch ihre Funktion ähnlich ist.
Im Jahr 1998 entdeckte man auch in der Pflanze Arabidopsis thaliana mögliche Glutamatrezeptorgene. Das war ein überraschender Fund, weil Pflanzen kein Nervensystem oder gar Gehirn haben. Dennoch besitzt Arabidopsis sogar zwei Glutamatrezeptorgene mehr als Wirbeltiere, wie sich nach der Sequenzierung ihres Erbgutes im Jahr 2000 herausstellte. Die Proteine, die von diesen Genen kodiert und als GLRs (glutamate-like receptors, glutamatähnliche Rezeptoren) bezeichnet werden, ähneln den Glutamatrezeptoren der Tiere sowohl in ihrer Sequenz als auch in ihrer vorhergesagten Struktur. Das legte die Vermutung nahe, daß auch ihre Funktion ähnlich ist.
 Die Funktion zu erforschen erwies sich jedoch als ungewöhnlich schwierig. Lange Zeit waren nur Analysen in der Pflanze erfolgreich, die es aber nicht erlaubten, die molekularen Eigenschaften einzelner GLR-Untereinheiten direkt zu charakterisieren. In einigen Fällen ließen sich die beobachteten Effekte nicht einmal mit letzter Sicherheit auf GLRs zurückführen. Insgesamt ergab sich ein sehr vielfältiges Bild möglicher Funktionen, darunter die Regulation des Kohlenstoff- und Stickstoffstoffwechsels, des Hormonhaushalts und der Wasser- und Ionenverteilung innerhalb der Pflanze sowie die Reaktion auf verschiedene Streßeinflüsse aus der Umwelt. Die Untersuchung einzelner, isolierter GLR-Proteine blieb dagegen lange erfolglos. Daher war unklar, ob sie tatsächlich ähnlich wie die Glutamatrezeptoren der Tiere funktionieren.
Die Funktion zu erforschen erwies sich jedoch als ungewöhnlich schwierig. Lange Zeit waren nur Analysen in der Pflanze erfolgreich, die es aber nicht erlaubten, die molekularen Eigenschaften einzelner GLR-Untereinheiten direkt zu charakterisieren. In einigen Fällen ließen sich die beobachteten Effekte nicht einmal mit letzter Sicherheit auf GLRs zurückführen. Insgesamt ergab sich ein sehr vielfältiges Bild möglicher Funktionen, darunter die Regulation des Kohlenstoff- und Stickstoffstoffwechsels, des Hormonhaushalts und der Wasser- und Ionenverteilung innerhalb der Pflanze sowie die Reaktion auf verschiedene Streßeinflüsse aus der Umwelt. Die Untersuchung einzelner, isolierter GLR-Proteine blieb dagegen lange erfolglos. Daher war unklar, ob sie tatsächlich ähnlich wie die Glutamatrezeptoren der Tiere funktionieren.
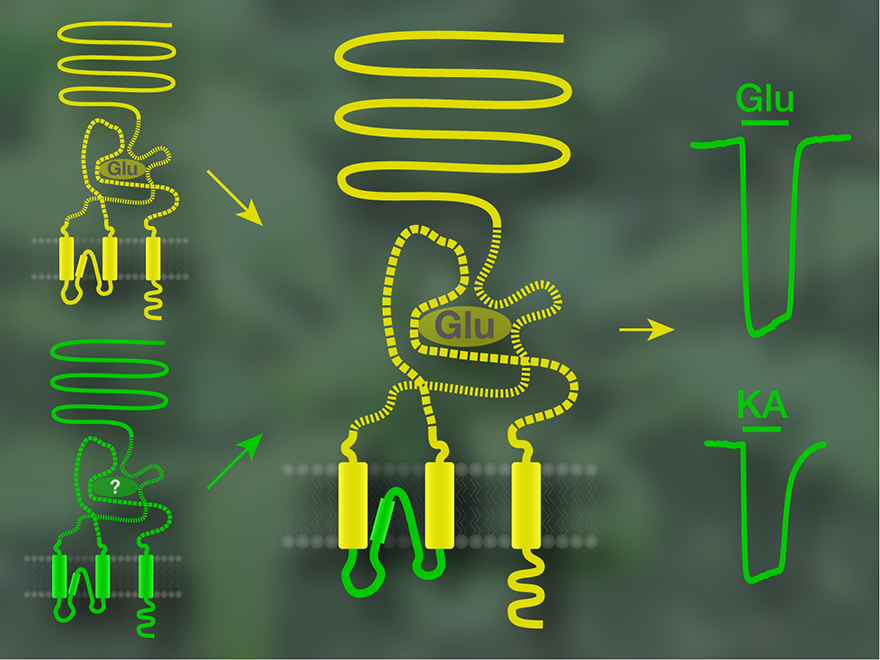 Im Jahr 2008 konnten wir nachweisen, daß einige GLR-Proteine tatsächlich einen funktionellen Ionenkanal besitzen. Dazu bauten wir den Teil des pflanzlichen Proteins, der vermutlich den Ionenkanal bildet, in einen tierischen Glutamatrezeptor anstelle seines eigenen Ionenkanals ein. Die Glutamatbindestelle blieb dabei erhalten, so daß wir den Rezeptor mit Glutamat aktivieren konnten. Auf diese Weise untersuchten wir die vermuteten Ionenkanalabschnitte von 17 GLR-Untereinheiten und erhielten in zwei Fällen einen chimären Rezeptor, der bei Aktivierung durch Glutamat Ionen in die Zelle strömen ließ. Damit war gezeigt, daß GLRs aus Arabidopsis prinzipiell Ionenkanäle bilden können.
Im Jahr 2008 konnten wir nachweisen, daß einige GLR-Proteine tatsächlich einen funktionellen Ionenkanal besitzen. Dazu bauten wir den Teil des pflanzlichen Proteins, der vermutlich den Ionenkanal bildet, in einen tierischen Glutamatrezeptor anstelle seines eigenen Ionenkanals ein. Die Glutamatbindestelle blieb dabei erhalten, so daß wir den Rezeptor mit Glutamat aktivieren konnten. Auf diese Weise untersuchten wir die vermuteten Ionenkanalabschnitte von 17 GLR-Untereinheiten und erhielten in zwei Fällen einen chimären Rezeptor, der bei Aktivierung durch Glutamat Ionen in die Zelle strömen ließ. Damit war gezeigt, daß GLRs aus Arabidopsis prinzipiell Ionenkanäle bilden können.
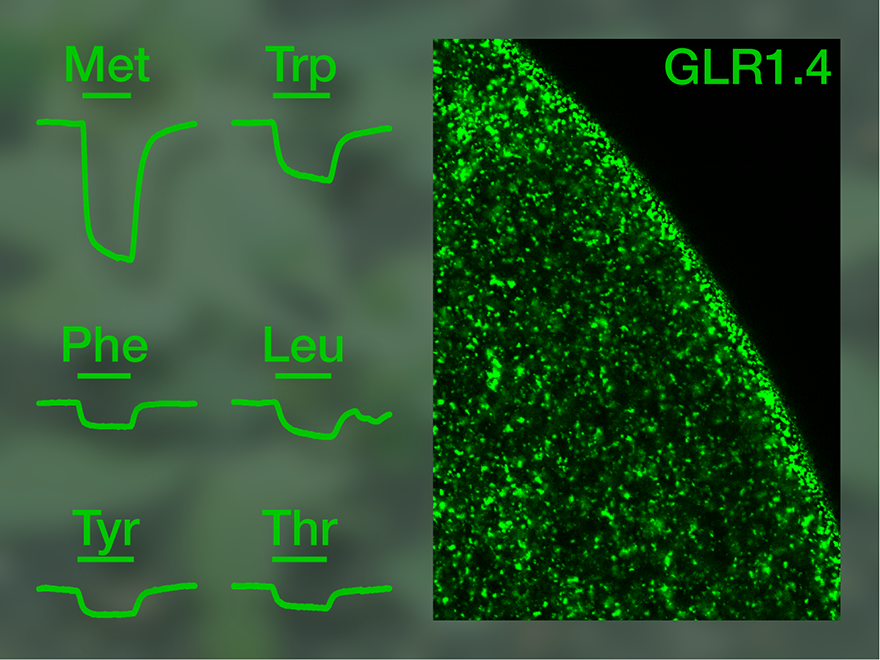 Im Anschluß konzentrierten wir uns auf die beiden GLR-Untereinheiten, bei denen wir in den Chimärenexperimenten einen funktionellen Ionenkanal identifiziert hatten. Die vollständigen pflanzlichen Rezeptoren reagierten nicht auf Glutamat, einer von ihnen ließ sich aber durch andere Aminosäuren so aktivieren, daß Ionen in die Zelle einströmten. Damit war nachgewiesen, daß zumindest diese GLR-Untereinheit, GLR1.4, ein ligandenaktivierter Ionenkanal ist, der ähnlich funktioniert wie die Glutamatrezeptoren aus Tieren. Eine ungewöhnliche Eigenschaft ist aber, daß nicht nur ein definierter Botenstoff den Rezeptor aktivieren kann, sondern mehrere verschiedene, aber miteinander verwandte Substanzen. Weil Glutamat unwirksam ist, kann GLR1.4 nicht als Glutamatrezeptor bezeichnet werden. Inzwischen wurden auch für eine weitere GLR-Untereinheit ähnliche Eigenschaften gefunden; auch sie ist ein Ionenkanal, der durch mehrere Aminosäuren aktiviert wird, allerdings durch andere als der von uns charakterisierte GLR1.4.
Im Anschluß konzentrierten wir uns auf die beiden GLR-Untereinheiten, bei denen wir in den Chimärenexperimenten einen funktionellen Ionenkanal identifiziert hatten. Die vollständigen pflanzlichen Rezeptoren reagierten nicht auf Glutamat, einer von ihnen ließ sich aber durch andere Aminosäuren so aktivieren, daß Ionen in die Zelle einströmten. Damit war nachgewiesen, daß zumindest diese GLR-Untereinheit, GLR1.4, ein ligandenaktivierter Ionenkanal ist, der ähnlich funktioniert wie die Glutamatrezeptoren aus Tieren. Eine ungewöhnliche Eigenschaft ist aber, daß nicht nur ein definierter Botenstoff den Rezeptor aktivieren kann, sondern mehrere verschiedene, aber miteinander verwandte Substanzen. Weil Glutamat unwirksam ist, kann GLR1.4 nicht als Glutamatrezeptor bezeichnet werden. Inzwischen wurden auch für eine weitere GLR-Untereinheit ähnliche Eigenschaften gefunden; auch sie ist ein Ionenkanal, der durch mehrere Aminosäuren aktiviert wird, allerdings durch andere als der von uns charakterisierte GLR1.4.
Die meisten pflanzlichen Glutamatrezeptorhomologe sind jedoch noch immer völlig uncharakterisiert, ihre Funktionen in der Pflanze sind weitgehend unklar, und auch die ungewöhnlichen Eigenschaften der bisher untersuchten Untereinheiten sind nicht im Detail verstanden. Wir arbeiten in unserer Gruppe daran, einige der offenen Fragen zu beantworten und damit zum besseren Verständnis der weiterhin ziemlich mysteriösen Familie der pflanzlichen GLR-Proteine beizutragen.
Eigene Publikationen zum Thema
- D. Tapken, U. Anschütz, L.-H. Liu, T. Huelsken, G. Seebohm, D. Becker, and M. Hollmann (2013).
A plant homolog of animal glutamate receptors is an ion channel gated by multiple hydrophobic amino acids.
Science Signaling 6(279): ra47.
doi: 10.1126/scisignal.2003762
Abstract Pressemitteilung

- D. Tapken and M. Hollmann (2008).
Arabidopsis thaliana glutamate receptor ion channel function demonstrated by ion pore transplantation.
Journal of Molecular Biology 383(1): 36-48.
doi: 10.1016/j.jmb.2008.06.076
Abstract
- D. Tapken and M. Hollmann (2006).
Homologs of mammalian glutamate receptors in invertebrates and plants.
In: Biological and biophysical aspects of ligand-gated ion channel receptor superfamilies, H. Arias, ed. Research Signpost, Trivandrum, pp. 321-382.
Abstract
- B. Lacombe, D. Becker, R. Hedrich, R. DeSalle, M. Hollmann, J.M. Kwak, J.I. Schröder, N. Le Novère, H.G. Nam, E.P. Spalding, M. Tester, F.J. Turano, J. Chiu, and G. Coruzzi (2001).
The identity of plant glutamate receptors.
Science 292(5521): 1486-1487.
doi: 10.1126/science.292.5521.1486b
Abstract
Doktorarbeiten zum Thema
- Thi Hong Thanh Lu
Untersuchungen zur Agonistenspezifität und Heteromerbildung von Arabidopsis-thaliana-Glutamatrezeptorhomologen (AtGLR)
September 2012 bis Dezember 2016
Arbeit als pdf herunterladen (34,8 MB)
- Daniel Tapken
Molekulare und funktionelle Charakterisierung pflanzlicher Glutamatrezeptoren
September 2002 bis August 2008
Arbeit als pdf herunterladen (21,7 MB)
Diplom- und Masterarbeiten zum Thema
- Franziska Schmoll
Untersuchung der Ligandenbindungsdomäne des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4 durch Punktmutagenese
bis 02.02.2021
Betreuer: Dr. Daniel Tapken
- Frederik Bodemer
Elektrophysiologische Untersuchung von Mutanten der Ligandenbindungsdomäne des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4
bis 21.08.2019
Betreuer: Dr. Daniel Tapken
- Andrea Mölders
Untersuchungen zur Rolle funktioneller Domänen pflanzlicher Glutamatrezeptorhomologe
bis 30.09.2011
Betreuer: Dr. Daniel Tapken
- Daniel Tapken
Untersuchung von Glutamatrezeptoren aus Arabidopsis thaliana
30.11.2001 bis 11.07.2002
Bachelorarbeiten zum Thema
- Ben Jakob Pahs
Untersuchung von Punktmutanten an der Dimergrenzfläche des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4
25.09.2025
Betreuer: Dr. Daniel Tapken
- Rowena Gänsler
Elektrophysiologische Untersuchung von pflanzlichen Glutamatrezeptorhomologen der Klade 3
05.10.2021
Betreuer: Dr. Daniel Tapken
- Laura Moreno Wasielewski
Untersuchung von Punktmutanten im M3-S2-Linker des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4
09.09.2021
Betreuer: Dr. Daniel Tapken
- Maria Jordan
Punktmutagenese im M3-S2-Linker des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4
21.09.2020
Betreuer: Dr. Daniel Tapken
- Janine Theile
Punktmutagenese in der S2-Domäne des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4
18.09.2020
Betreuer: Dr. Daniel Tapken
- N.N.
Charakterisierung von Chimären in der Linkerregion des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4
11.09.2019
Betreuer: Dr. Daniel Tapken
- Sebastian Mesch
Mutagenese in der Linkerregion des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4
25.09.2018
Betreuer: Dr. Daniel Tapken
- Jil Schrader
Punktmutation in der Porenhelix des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4
28.09.2017
Betreuer: Dr. Daniel Tapken
- Klara Scholtissek
Mutagenese in der Ligandenbindungsdomäne des pflanzlichen Glutamatrezeptorhomologs AtGLR1.4
28.09.2017
Betreuer: Dr. Daniel Tapken
- Bilhan Karacora
Herstellung und Charakterisierung von Chimären innerhalb der Klade 1 pflanzlicher Glutamatrezeptorhomologe
27.09.2016
Betreuer: Dr. Daniel Tapken
- Marvin Mann
Erzeugung und Charakterisierung von Porenchimären pflanzlicher Glutamatrezeptorhomologe
29.05.2015
Betreuer: Dr. Daniel Tapken
- Karolina Kleinsimlinghaus
Punktmutagenese in der Porenregion eines pflanzlichen Glutamatrezeptorhomologs
06.10.2010
Betreuer: Dr. Daniel Tapken
- Sonja Kühn
Konstruktion fluoreszenzmarkierter Arabidopsis thaliana-Glutamatrezeptoren
07.11.2005
Betreuer: Daniel Tapken



 Amina Bećić
Amina Bećić Franziska Schmoll
Franziska Schmoll Frederik Bodemer
Frederik Bodemer Dr. Thi Hong Thanh Lu
Dr. Thi Hong Thanh Lu Andrea Mölders
Andrea Mölders